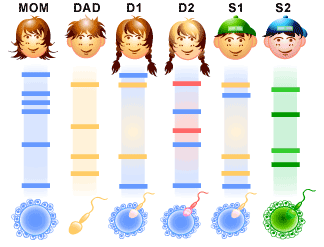
9ª postagem: DNA FINGERPRINT E "IDENTIDADE MOLECULAR"
 |
| DNA fingerprint e a "identidade molecular" |
“DNA
Fingerprints” têm sido utilizados para descrever o padrão molecular de um
genótipo. Eles podem ser obtidos a partir de vários tipos de marcadores moleculares.
Resumidamente estes marcadores moleculares podem ser classificados em dois
grupos, a depender da metodologia utilizada para identificá-los:
 |
| DNA fingerprint e os Testes de Paternidade |
 |
| DNA fingerprint e identificação de suspeitos |
1)
hibridização de DNA (método RFLP: Polimorfismo de comprimento de fragmentos de restrição);
2) amplificação de DNA (RAPD: DNA polimórfico amplificado aleatoriamente).
Para cada tipo de marcador de DNA existem
vantagens e limitações.
O método por hibridização do DNA, RFLP, vai utilizar dos diferentes padrões nas sequências dos nucleotídeos do DNA. As enzimas de restrição vão reconhecer sequências específicas, que em DNAs diferentes podem estar em posições diferentes, e vão cortá-las, gerando fragmentos de DNA com pesos moleculares diferentes. Esses fragmentos são diferenciados através de uma eletroforese.
A dificuldade desse método está no fato de que, como muitas sequências se repetem bastante no DNA, o uso dessa técnica provocará um número enorme de fragmentos, impedindo a distinção por visualização depois da eletroforese.
Para reduzir essa dificuldade, é usada uma prova de identificação. Os protocolos determinam que se usem 7 alelos de baixa ocorrência dentro de uma população (mas esse número pode variar. O FBI, por exemplo, usa 13 alelos). O uso de sondas para reconhecimento desses alelos e posterior sensibilização de filme radiográfico vai permitir uma distinção mais clara dos fragmentos de DNA obtidos.
 |
| Método RFLP de identificação da Impressão Digital de DNA |
Já o método por amplificação do DNA, RAPD, utiliza o método da Reação em Cadeia da Polimerase. Primers pequenos são desenhados e colocados para hibridizar o DNA aleatoriamente. Se forem usados 7 pares de primers, serão necessárias 7 reações. Posteriormente à PCR, os produtos terão tamanhos diferenciados, que são distinguidos na eletroforese.
A
facilidade, rapidez, versatilidade e sensibilidade da PCR, a torna particularmente
poderosa para estudos genético-moleculares envolvendo grande número de
indivíduos de qualquer organismo vivo. Muitos métodos tradicionais de clonagem,
sequenciamento e análise de polimorfismo de DNA foram acelerados ou
substituídos pelo uso das inúmeras derivações da técnica de PCR.
A
técnica de PCR utilizando iniciadores de sequência arbitrária abriu uma
perspectiva inteiramente nova para a análise genômica de indivíduos e populações.
Além de facilitar e acelerar os estudos que já ocorriam com as espécies mais
tradicionais, a tecnologia RAPD trouxe uma verdadeira “democratização” da
análise de polimorfismo molecular, ao permitir a realização de estudos de
análise genética em espécies anteriormente não contempladas.
 |
| PCR e a técnica RAPD |
O
uso da técnica RAPD não requer experiência aprofundada em biologia molecular e
nem tampouco instalações sofisticadas de laboratório. É uma tecnologia bastante
accessível, que pode ser transferida diretamente para estações experimentais de
melhoramento, para laboratórios avançados de pesquisa a ser utilizada
cotidianamente pelo geneticista.
A
principal limitação dos marcadores RAPD é o baixo conteúdo de informação
genética por loco. Apenas um alelo é detectado: o segmento que é amplificado,
enquanto que as demais variações alélicas são classificadas conjuntamente com
um alelo nulo.
 |
| Padrão obtido depois de uma corrida eletroforética |
Fontes: Aula de "Impressão Digital de DNA" do professor Fernando Aécio
http://uenf.br/pos-graduacao/gmp/files/2012/01/Tese-MS-Carolina-M-Palacios-de-Souza.pdf