9ª postagem: DNA FINGERPRINT E "IDENTIDADE MOLECULAR"
 |
| DNA fingerprint e a "identidade molecular" |
“DNA
Fingerprints” têm sido utilizados para descrever o padrão molecular de um
genótipo. Eles podem ser obtidos a partir de vários tipos de marcadores moleculares.
Resumidamente estes marcadores moleculares podem ser classificados em dois
grupos, a depender da metodologia utilizada para identificá-los:
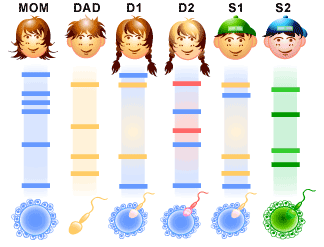 |
| DNA fingerprint e os Testes de Paternidade |
 |
| DNA fingerprint e identificação de suspeitos |
1)
hibridização de DNA (método RFLP: Polimorfismo de comprimento de fragmentos de restrição);
2) amplificação de DNA (RAPD: DNA polimórfico amplificado aleatoriamente).
Para cada tipo de marcador de DNA existem
vantagens e limitações.
O método por hibridização do DNA, RFLP, vai utilizar dos diferentes padrões nas sequências dos nucleotídeos do DNA. As enzimas de restrição vão reconhecer sequências específicas, que em DNAs diferentes podem estar em posições diferentes, e vão cortá-las, gerando fragmentos de DNA com pesos moleculares diferentes. Esses fragmentos são diferenciados através de uma eletroforese.
A dificuldade desse método está no fato de que, como muitas sequências se repetem bastante no DNA, o uso dessa técnica provocará um número enorme de fragmentos, impedindo a distinção por visualização depois da eletroforese.
Para reduzir essa dificuldade, é usada uma prova de identificação. Os protocolos determinam que se usem 7 alelos de baixa ocorrência dentro de uma população (mas esse número pode variar. O FBI, por exemplo, usa 13 alelos). O uso de sondas para reconhecimento desses alelos e posterior sensibilização de filme radiográfico vai permitir uma distinção mais clara dos fragmentos de DNA obtidos.
 |
| Método RFLP de identificação da Impressão Digital de DNA |
Já o método por amplificação do DNA, RAPD, utiliza o método da Reação em Cadeia da Polimerase. Primers pequenos são desenhados e colocados para hibridizar o DNA aleatoriamente. Se forem usados 7 pares de primers, serão necessárias 7 reações. Posteriormente à PCR, os produtos terão tamanhos diferenciados, que são distinguidos na eletroforese.
A
facilidade, rapidez, versatilidade e sensibilidade da PCR, a torna particularmente
poderosa para estudos genético-moleculares envolvendo grande número de
indivíduos de qualquer organismo vivo. Muitos métodos tradicionais de clonagem,
sequenciamento e análise de polimorfismo de DNA foram acelerados ou
substituídos pelo uso das inúmeras derivações da técnica de PCR.
A
técnica de PCR utilizando iniciadores de sequência arbitrária abriu uma
perspectiva inteiramente nova para a análise genômica de indivíduos e populações.
Além de facilitar e acelerar os estudos que já ocorriam com as espécies mais
tradicionais, a tecnologia RAPD trouxe uma verdadeira “democratização” da
análise de polimorfismo molecular, ao permitir a realização de estudos de
análise genética em espécies anteriormente não contempladas.
 |
| PCR e a técnica RAPD |
O
uso da técnica RAPD não requer experiência aprofundada em biologia molecular e
nem tampouco instalações sofisticadas de laboratório. É uma tecnologia bastante
accessível, que pode ser transferida diretamente para estações experimentais de
melhoramento, para laboratórios avançados de pesquisa a ser utilizada
cotidianamente pelo geneticista.
A
principal limitação dos marcadores RAPD é o baixo conteúdo de informação
genética por loco. Apenas um alelo é detectado: o segmento que é amplificado,
enquanto que as demais variações alélicas são classificadas conjuntamente com
um alelo nulo.
 |
| Padrão obtido depois de uma corrida eletroforética |
Fontes: Aula de "Impressão Digital de DNA" do professor Fernando Aécio
http://uenf.br/pos-graduacao/gmp/files/2012/01/Tese-MS-Carolina-M-Palacios-de-Souza.pdf
É importante saber que se dois organismos diferirem na distância entre os sítios de clivagem de uma endonuclease de restrição, o comprimento dos fragmentos produzidos vai diferir quando o DNA for digerido com uma enzima de restrição.
ResponderExcluirPara a detecção de RFLPs pode ser usada a metodologia de Southern Blot, que permite a identificação de fragmentos de DNAcom sequências idênticas ou similares à sonda utilizada e pode ajudar no posicionamento relativo de diferente fragmentos dentro de um fragmento maior de DNA
FONTE:
http://publicacoes.unigranrio.edu.br/index.php/sare/article/viewFile/242/231
Apesar de a maioria das sequências de DNA humano serem comuns em humanos, a impressão genética faz uso de sequências repetitivas altamente variáveis, chamadas VNTR (variable number tandem repeats). Estes loci são altamente similares entre humanos muito relacionados, mas variáveis o suficiente para que seja extremamente pouco comum humanos pouco relacionados terem os mesmos alelos. Esta técnica foi primeiramente reportada em 1985 por Sir Alec Jeffreys na Universidade de Leicester, na Inglaterra, e é agora a base de várias bases de dados nacionais.
ResponderExcluirReferências:
http://bgdsancho1.no.sapo.pt/doc_ampl/03_DNA_fingerprint.htm
Já que, como dito na postagem, a técnica de DNA fingerprint tem o poder de ser usada em resolução de casos emblemáticos e importantes ela esbarra em questões jurídicas. Como explicado, essa técnica é usada na identificação de indivíduos, assim como um "código de barras". No entanto, ela não está isenta de erros, principalmente erros humanos de aplicação. Para que haja credibilidade dos exames de DNA é necessário, ao longo das várias fases de execução da tipagem por DNA, aplicar rigorosos procedimentos para a garantia da qualidade dos serviços periciais. A omissão na aplicação dos controles de qualidade efetivamente pode levar a interpretação equivocada dos resultados. Em investigações genéticas, a validade dos resultados alcançados depende do cálculo das frequências populacionais dos marcadores empregados. Para isto, as fontes destas frequências devem estar disponíveis a qualquer pessoa relacionada ao processo que necessite desta informação. Pode-se encontrar significativas variações na composição genica entre grupos populacionais, sendo este um fator importante a ser considerado.
ResponderExcluirFonte: http://www.ambito-juridico.com.br/site/index.php?artigo_id=1175&n_link=revista_artigos_leitura
Além das técnicas de RFLP e RAPD apontadas na postagem, destacam-se também dois outros tipos de marcadores moleculares: o AFLP e os microssatélites. Os marcadores gerados pela análise de polimorfismos de comprimento de fragmento amplificado (AFLP) associam os polimorfismos gerados por enzimas de restrição com a capacidade de detecção da técnica de PCR. O DNA total é clivado por enzimas de restrição, originando um número extremamente elevado de fragmentos que, em função da concentração, não são detectados em eletroforese. Pequenas sequências de DNA (adaptadores) são acopladas às extremidades desses fragmentos de restrição, as quais se anelarão com primers específicos, durante a PCR. Os fragmentos gerados são então separados por eletroforese em gel de poliacrilamida desnaturante. A técnica de AFLP apresenta vantagens comparativas, tais como detecção de maior número de lócus, cobertura ampla do genoma e baixo custo. O DNA microssatélite consiste de unidades de cerca de 1 a 6 nucleotídeos repetidas em tandem (em sequência) dentro do genoma de vários organismos, pricipalmente eucariotos. Os microsatélites são utilizados como marcadores moleculares devido ao alto nível de polimorfismo encontrado em seus locos, o que proporciona sua utilização em diversos propósitos de estudo populacional, permitindo analisar desde indivíduos até espécies proximamente relacionadas. O deslizamento (slippage) da DNA polimerase durante a replicação do DNA é tida como a principal causa da variação no número de repetições nesses locos. Os diferentes números de repetições caracterizam os diferentes alelos, que podem ser detectados numa eletroforese em gel de poliacrilamida. Como o DNA microssatélite normalmente não carrega informação, as adições ou deleções de unidades repetitivas não são selecionadas, o que proporciona alta variabilidade de alelos.
ResponderExcluirFontes: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-29452005000300027
http://www2.bioqmed.ufrj.br/prosdocimi/chicopros/ensino/didaticos/microsatelites.html
Outra aplicação importante do método RFLP além da medicina forense é no diagnóstico de doenças de caráter genético. Alguns exemplos são a Fibrose Cística (FC) e a Anemia Falciforme (AF). No caso da FC, ela se trata de uma doença recessiva, o que quer dizer que para expressar o seu fenótipo, o paciente deve ser homozigótico recessivo para os alelos da doença. É fácil, portanto, através da comparação, definir se a pessoa é homozigótica normal (saudável), heterozigótica (apenas portadora da doença) ou homozigótica afetada (porta a doença e a exprime). Já no caso da anemia falciforme, ambos os genes no paciente substituem um T por um A na posição do meio do codão 6. Isto converte o codão GAG (para o glutamato) ao codão GTG (para a valina) e elimina uma sequência reconhecida (CTGAGG que é comum aos codões 5, 6 e 7). Quando o gene normal (beta a), que possui o codão GAG, é digerido com uma enzima de restrição e os fragmentos são separados por electroforese a sonda liga-se a um pequeno fragmento (entre as setas vermelhas). Quando é o gene doente (beta s), com o codão GTG, a enzima não pode cortá-lo neste sítio, então a sonda liga-se a um fragmento muito maior. Essa diferença de tamanho que gera o polimorfismo observado na eletroforese.
ResponderExcluirFonte: http://www.ebah.com.br/content/ABAAAAjM4AB/tecnicas-analise-dna
O grande avanço na área de marcadores moleculares baseados em PCR com a idéia de utilizar primers mais curtos e de sequência arbitrária para dirigir a reação de amplificação, eliminando assim a necessidade do conhecimento prévio de sequência gênica, possibilitando o desenvolvimento da técnica de RAPD como foi exeplificada na postagem. Além de facilitar a obtenção de “fingerprints” genômicos de indivíduos, variedades e populações, a tecnologia RAPD permitiu a realização de estudos em espécies anteriormente não contempladas. As aplicações incluem: Acelerar os estudos que já ocorriam com as espécies tradicionais (ex: milho, tomate, arroz); Análise da estrutura e diversidade genética em populações naturais, populações de melhoramento e bancos de germoplasma; Estabelecimento de relacionamentos filogenéticos entre diferentes espécies; Construção de mapas genéticos de alta cobertura genômica e a localização de genes de interesse econômico.
ResponderExcluirFonte: http://geneticavirtual.webnode.com.br/genetica-virtual-home/topicos-extras/marcadores-moleculares/polimorfismo-de-dna-amplificado-ao-acaso-rapd/
Pessoas diferentes podem ter números diferentes de repetições destas sequências: uma pessoa pode ter cinco repetições num locus (local) de DNA específico; outra pessoa pode ter sete. Utilizando amostras, por exemplo de sangue ou sémen, pode-se analisar as sequências repetitivas em diversos loci de DNA; chama-se a esta análise uma impressão digital genética. Tal como as impressões digitais, as impressões digitais genéticas podem ser usadas para distinguir indivíduos.
ResponderExcluirApesar do termo “impressão digital genética” (ou perfil genético) ser habitualmente usado, nem todos estão cientes de que na verdade esta corresponde a duas técnicas muito diferentes, apenas uma das quais é frequentemente usada nas ciências forenses atualmente.
http://www.scienceinschool.org/2012/issue22/fingerprinting/portuguese
A técnica de RAPD, citada no texto, é basicamente uma variação do protocolo de PCR, com duas características distintas: utiliza um primer único ao invés de um par de primers; o primer único tem sequência arbitrária, e portanto sua sequência alvo é desconhecida.Para que haja amplificação de um fragmento RAPD no genoma, duas sequências de DNA complementares ao primer arbitrário devem estar suficientemente adjacentes (< 4000 pares de bases) e em orientação oposta, de maneira a permitir a amplificação exponencial de um segmento de DNA pela DNA polimerase. Em função da grande quantidade de DNA produzido, este segmento pode ser visualizado na forma de banda num gel de eletroforese. Cada primer arbitrário dirige a síntese de vários segmentos de DNA simultaneamente em diversos pontos do genoma, resultando em várias bandas no gel.
ResponderExcluirFonte: http://geneticavirtual.webnode.com.br/genetica-virtual-home/topicos-extras/marcadores-moleculares/polimorfismo-de-dna-amplificado-ao-acaso-rapd/